では、実際の実験室で行われているゲノム編集に関して記述してみます。
必要な「部品」は
① Cas9 発現ベクター
② sgRNA 発現ベクター
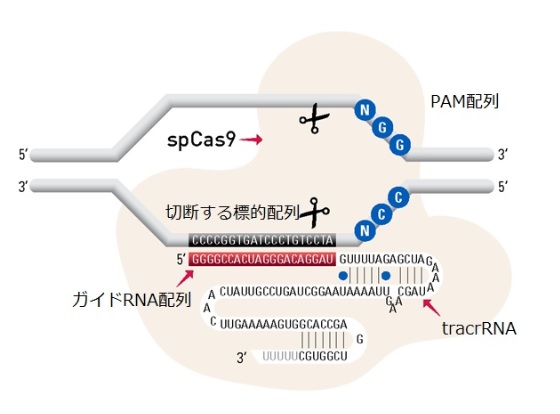
の2種類だけです。(CRISPR/Cas9_1 で解説した tracrRNA と crRNA は一本化されて sgRNA となっています)
この2種類のベクターを細胞に導入(トランスフェクション)するだけです。この2種のベクターを取り込んだ細胞では、CRISPR/Cas9 が狙った DNA 配列を切断してくれます。
Cas9 発現ベクターは市販されているので、それを購入することになります。狙った遺伝子配列に対する sgRNA 発現ベクターを作ることが研究者の「仕事」になります。
(、、、が、もし、あなたの研究室に金銭的な余裕があれば、、、sgRNA 発現ベクターを作成してくれる会社があるので、そこに「狙う DNA 配列」を記載して申し込むと、数週間でベクターを作ってくれます)
フナコシ株式会社のホーム・ページ「ゲノム編集ツール CRISPR-Cas9 システム特集 」より https://www.funakoshi.co.jp/contents/8508
sgRNA 発現ベクターを自作するには
①狙う DNA 配列を決める。PAM 配列*に注意する。
② PCR法で「狙うDNA配列」を含んだ配列を増幅する。
③ PCR産物をベクターに組み込む。
という作業を行います。
sgRNA 発現ベクター用のPCR産物の構造は 5' 側から順に
1.U6 プロモーター配列(H1プロモーター配列でも良い)
2.狙ったDNA 配列
3.Scaffold 配列(CRISPR/Cas9_1 で解説した crRNA の「狙ったDNA配列」とは関係のない配列とtracrRNA
配列を結合した配列)
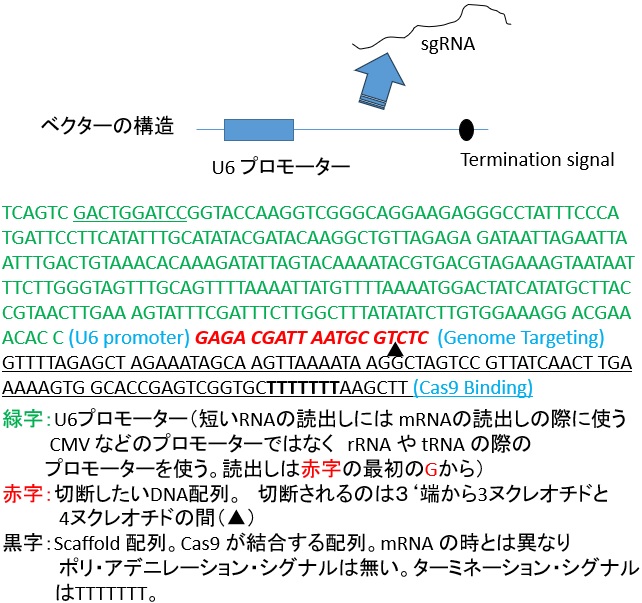
U6配列とScaffold 配列は、常に同一の DNA 配列なので、中央の「狙ったDNA配列」だけ、オリゴヌクレオチドとして合成して、PCR法のプライマーとし、PCR産物を合成します。
*PAM配列:狙ったDNA配列の3'末端には NGG という配列(PAM配列)がなければなりません。

